Single (i) Cell R 패키지 (iCellR)
iCellR은 고처리량 단일 세포 시퀀싱 데이터를 분석하고 시각화하는 데 도움을 주기 위해 설계된 인터랙티브 R 패키지입니다.scRNA-Seq, scVDJ-Seq, scATAC-Seq, CITE-Seq, 그리고 Spatial Transcriptomics (ST) 등 다양한 단일 세포 기술을 지원합니다.유지관리자: Alireza Khodadadi-Jamayran
뉴스 (2021년 4월)
scATAC-seq 및 Spatial Transcriptomics (ST) 분석을 위해 최신 버전 iCellR (v1.6.4)을 사용하세요.Tirosh, Mean, Sum, GSVA, ssgsea, Zscore, Plage와 같은 방법을 활용하여 유전자 서명 기반 세포 점수 매기기를 위한 i.score 기능을 활용하세요.뉴스 (2020년 7월)
세포 주기 분석 도구(단계 G0, G1S, G2M, M, G1M, S)를 포함한 iCellR 버전 1.5.5를 탐색하세요. 예제 phase를 확인하고, 새로운 의사시간 추상 KNetL (PAK map) 기능이 추가되어 의사시간 진행 상황을 시각화할 수 있습니다 (PAK map). 업데이트된 시각화 도구를 사용해 유전자 간 상관관계 분석을 수행하세요. correlations.뉴스 (2020년 5월)
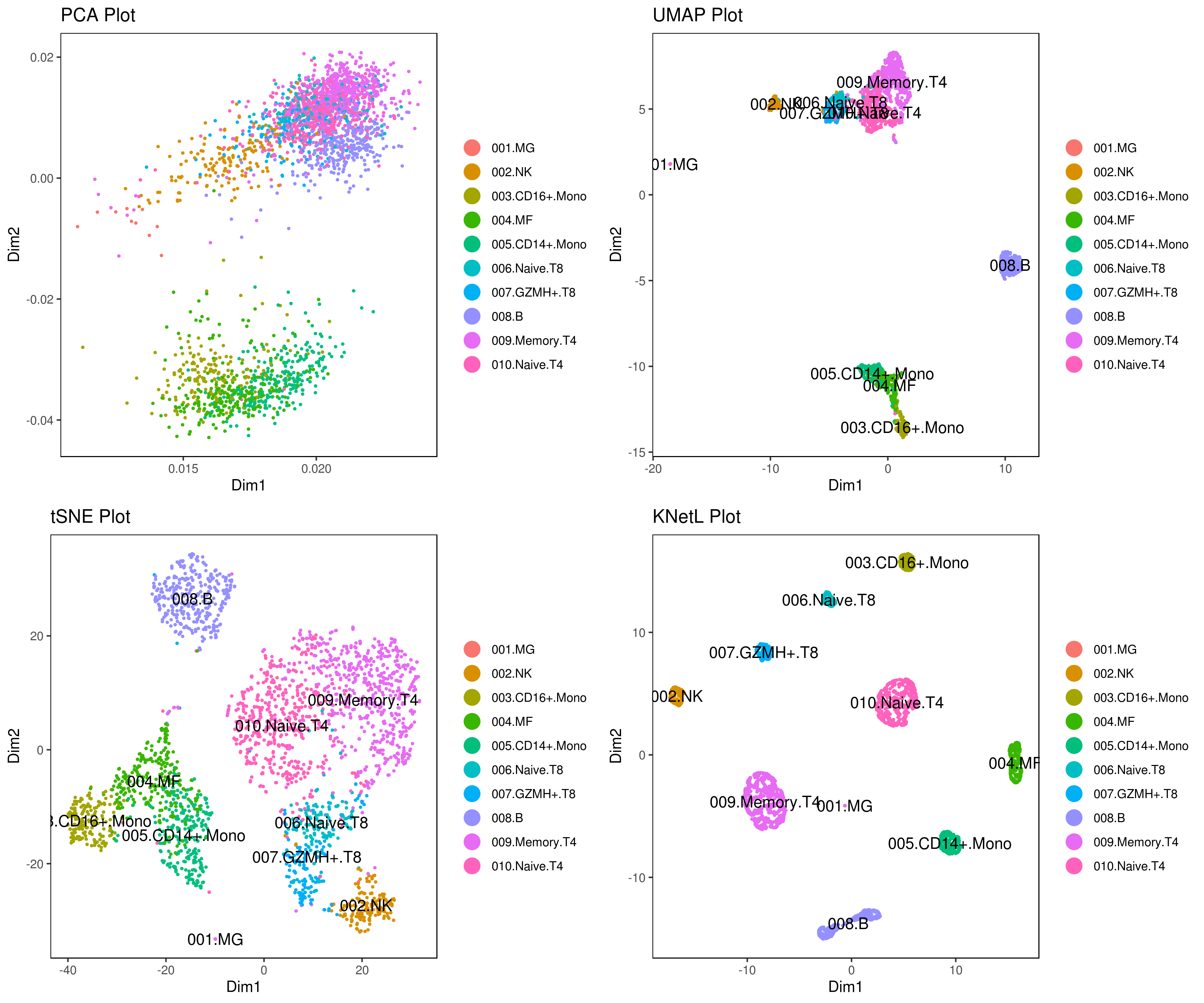
고급 조정 가능하고 동적인 차원 축소 방법인KNetL map을 탐색하세요 KNetL map 뉴스 (2020년 4월)
유전자 간 상관관계 분석을 개선하기 위한보간 및 커버리지 보정 (CC) 방법을 도입합니다. (CC). iCellR의 CPCA 및 CCCA 도구를 사용한 배치 정렬 수행 (CCCA 및 CPCA) 방법. 세포 유형 예측을 위한 데이터베이스가 ImmGen 및 MCA를 포함하여 확장되었습니다.뉴스 (2018년 9월)
scSeqR가 iCellR로 이름이 변경되었으며 scSeqR는 더 이상 지원되지 않습니다. 앞으로 iCellR을 사용하세요. UMAP이 iCellR에 추가되었습니다. 인터랙티브 세포 게이팅 기능이 추가되어 Plotly를 사용한 HTML 플롯 내에서 직접 세포를 선택할 수 있습니다.튜토리얼 및 매뉴얼
매뉴얼링크 Manual 및 Comprehensive R Archive Network (CRAN).시작하기및튜토리얼은 Wiki 페이지를 방문하세요.- CITE-Seq 및 scRNA-Seq 분석을 위한 비디오 튜토리얼 링크: Video
- KNetL map에 관한 모든 것: Video
FlowJo또는SeqGeq를 사용하는 경우 iCellR 및 기타 단일 세포 분석 도구용 플러그인을 제공합니다. 모든 플러그인 목록은 여기에서 확인할 수 있습니다: https://www.flowjo.com/exchange/#/ . 특히 iCellR 플러그인은 여기에서 찾을 수 있습니다: https://www.flowjo.com/exchange/#/plugin/profile?id=34. 또한 SeqGeq 차등 발현(DE) 튜토리얼이 제공되어 과정을 안내합니다: SeqGeq DE tutorial
iCellR 인용 시에는 이 PMID: 34353854를 사용하세요.iCellR publications: PMID: 35660135 (scRNA-seq/KNetL) PMID: 35180378 (CITE-seq/KNetL), PMID: 34911733 (i.score and cell ranking), PMID: 3496305501427-6?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0092867421014276%3Fshowall%3Dtrue) (scRNA-seq), PMID 31744829 (scRNA-seq), PMID: 31934613 (bulk RNA-seq from TCGA), PMID: 32550269 (scVDJ-seq), PMID: 34135081, PMID: 33593073, PMID: 34634466, PMID: 35302059, PMID: 34353854
Single (i) Cell R package (iCellR)






*
For getting started and tutorials go to our Wiki page.
--- Tranlated By Open Ai Tx | Last indexed: 2025-12-16 ---
