Paquet R Single (i) Cell (iCellR)
iCellR est un paquet R interactif conçu pour faciliter l'analyse et la visualisation des données de séquençage à cellule unique à haut débit. Il supporte diverses technologies de cellule unique, y comprisscRNA-Seq, scVDJ-Seq, scATAC-Seq, CITE-Seq et Transcriptomique Spatiale (ST).Mainteneur : Alireza Khodadadi-Jamayran
Actualités (avril 2021)
Utilisez la dernière version d’iCellR (v1.6.4) pour les analyses scATAC-seq et Transcriptomique Spatiale (ST). Profitez de la fonction i.score pour lescorage des cellules basé sur des signatures géniques avec des méthodes telles que Tirosh, Mean, Sum, GSVA, ssgsea, Zscore et Plage.Actualités (juillet 2020)
Découvrez iCellR version 1.5.5, désormais doté d’outils pour l’analyse du cycle cellulaire(phases G0, G1S, G2M, M, G1M et S). Voir l’exemple phase, nouvelle fonctionnalité Pseudotime Abstract KNetL (carte PAK) ajoutée – visualisez la progression du pseudotemps (carte PAK). Effectuez une analyse de corrélation gène-gène avec des outils de visualisation mis à jour. corrélations.Actualités (mai 2020)
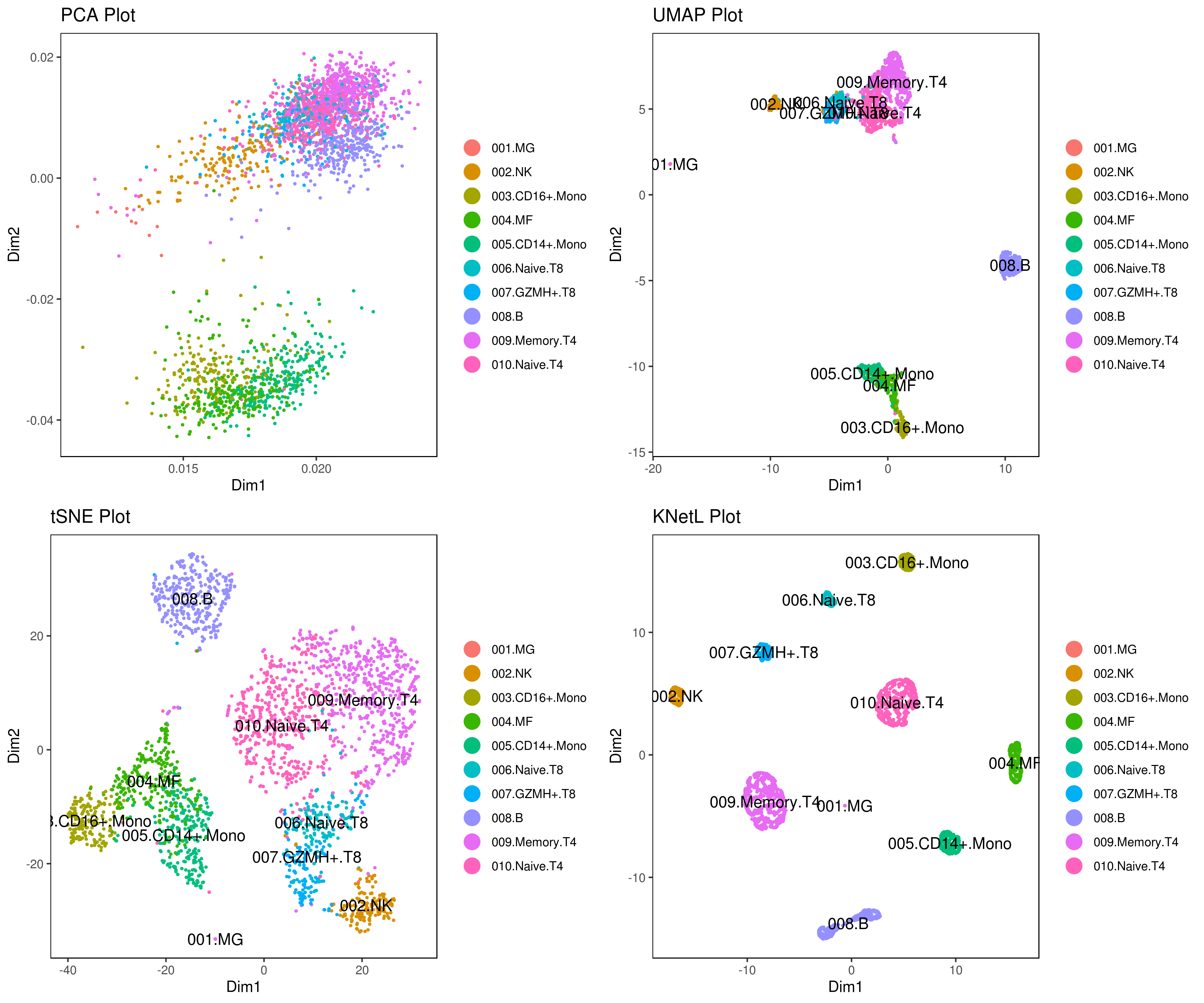
Explorez lacarte KNetL, une méthode avancée de réduction dimensionnelle ajustable et dynamique carte KNetL Actualités (avril 2020)
Introduction des méthodesimputation et correction de couverture (CC) pour une meilleure analyse de corrélation gène-gène. (CC). Effectuez un alignement de lot avec les outils CPCA et CCCA d’iCellR (CCCA et CPCA) méthodes. Bases de données élargies pour la prédiction du type cellulaire incluant désormais ImmGen et MCA.Actualités (sept. 2018)
scSeqR a été renommé en iCellR, et scSeqR a été abandonné. Merci d’utiliser iCellR dorénavant, car scSeqR n’est plus supporté. UMAP est ajouté à iCellR. Le gating cellulaire interactif a été ajouté, permettant aux utilisateurs de sélectionner des cellules directement dans les graphiques HTML via Plotly.Tutoriels et manuel
- Lien vers le
manuelManuel et Comprehensive R Archive Network (CRAN). - Pour
démarreret accéder auxtutoriels, rendez-vous sur notre page Wiki. - Lien vers un tutoriel vidéo pour l’analyse CITE-Seq et scRNA-Seq : Vidéo
- Tout ce que vous devez savoir sur la carte KNetL : Vidéo
- Si vous utilisez
FlowJoouSeqGeq, ils proposent des plugins pour iCellR et d’autres outils d’analyse à cellule unique. Vous pouvez trouver la liste complète des plugins ici : https://www.flowjo.com/exchange/#/ . Plus précisément, le plugin iCellR est disponible ici : https://www.flowjo.com/exchange/#/plugin/profile?id=34. De plus, un tutoriel SeqGeq sur l’Expression Différentielle (DE) est disponible pour vous guider : Tutoriel SeqGeq DE
citer iCellR, utilisez ce PMID : 34353854iCellR publications: PMID: 35660135 (scRNA-seq/KNetL) PMID: 35180378 (CITE-seq/KNetL), PMID: 34911733 (i.score and cell ranking), PMID: 3496305501427-6?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0092867421014276%3Fshowall%3Dtrue) (scRNA-seq), PMID 31744829 (scRNA-seq), PMID: 31934613 (bulk RNA-seq from TCGA), PMID: 32550269 (scVDJ-seq), PMID: 34135081, PMID: 33593073, PMID: 34634466, PMID: 35302059, PMID: 34353854
Single (i) Cell R package (iCellR)






*
For getting started and tutorials go to our Wiki page.
--- Tranlated By Open Ai Tx | Last indexed: 2025-12-16 ---
