Paquete R para célula única (i) (iCellR)
iCellR es un paquete interactivo de R diseñado para facilitar el análisis y la visualización de datos de secuenciación unicelular de alto rendimiento. Soporta una variedad de tecnologías unicelulares, incluyendoscRNA-Seq, scVDJ-Seq, scATAC-Seq, CITE-Seq y Transcriptómica Espacial (ST).Mantenedor: Alireza Khodadadi-Jamayran
Noticias (abril 2021)
Use la última versión de iCellR (v1.6.4) para análisis de scATAC-seq y Transcriptómica Espacial (ST). Aproveche la función i.score parapuntuar células basándose en firmas génicas usando métodos como Tirosh, Media, Suma, GSVA, ssgsea, Zscore y Plage.Noticias (julio 2020)
Explore la versión 1.5.5 de iCellR, ahora con herramientas para análisis del ciclo celular(fases G0, G1S, G2M, M, G1M y S). Vea un ejemplo de fase, nueva funcionalidad de Pseudotiempo Abstracto KNetL (mapa PAK) añadida – visualice la progresión del pseudotiempo (mapa PAK). Realice análisis de correlación gen-gen usando herramientas de visualización actualizadas. correlaciones.Noticias (mayo 2020)
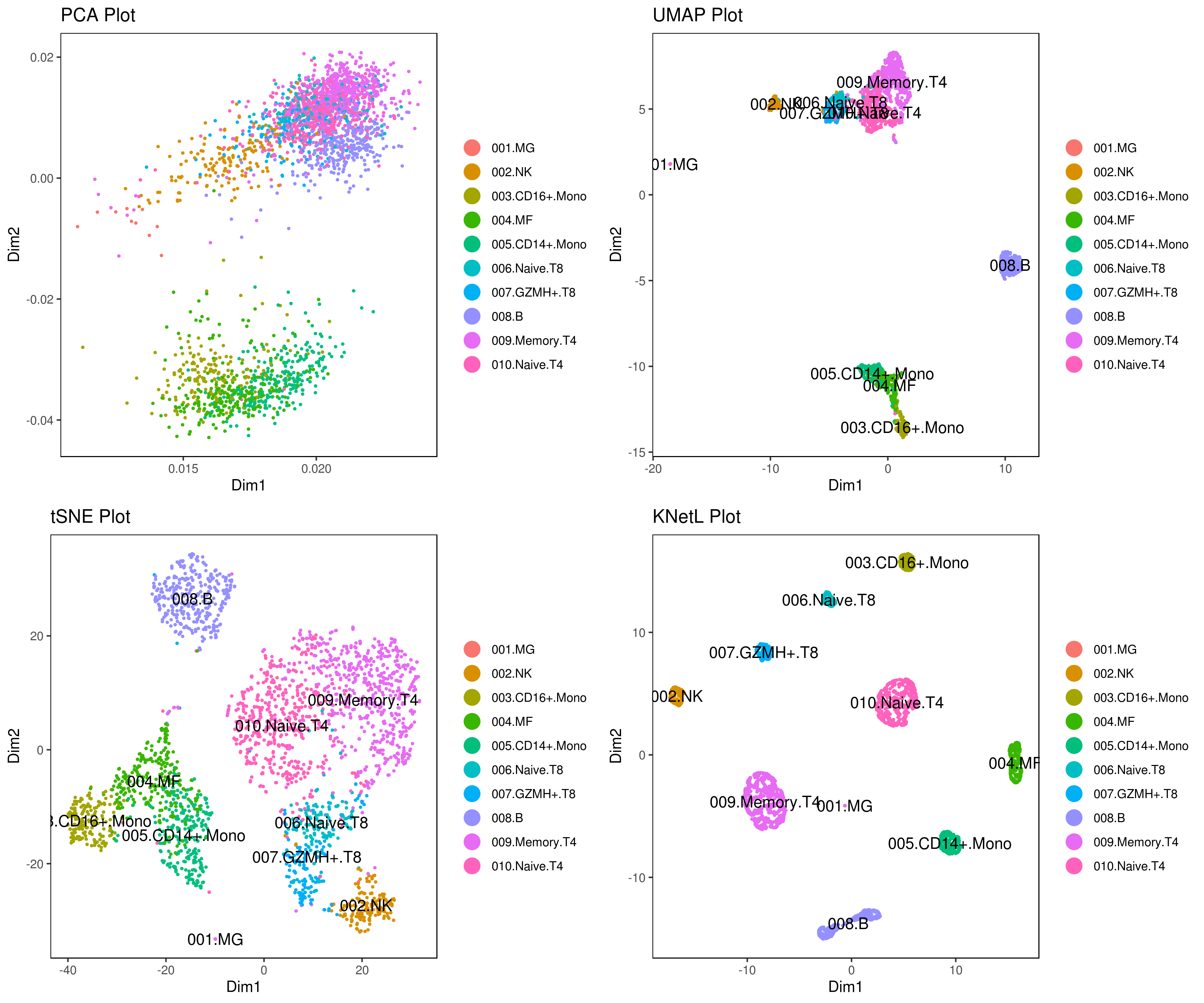
Explore elmapa KNetL, un método avanzado, ajustable y dinámico de reducción dimensional mapa KNetL Noticias (abril 2020)
Introducción de métodos deimputación y corrección de cobertura (CC) para mejorar el análisis de correlación gen-gen. (CC). Realice alineación de lotes usando CPCA y herramientas CCCA de iCellR (CCCA y CPCA) métodos. Bases de datos ampliadas para predicción de tipos celulares que ahora incluyen ImmGen y MCA.Noticias (sep. 2018)
scSeqR ha sido renombrado a iCellR, y scSeqR ha sido descontinuado. Por favor utilice iCellR en adelante, ya que scSeqR ya no tiene soporte. UMAP se añade a iCellR. Se ha incorporado selección interactiva de células, permitiendo a los usuarios seleccionar células directamente dentro de gráficos HTML usando Plotly.Tutoriales y manual
- Enlace al
manualManual y al Comprehensive R Archive Network (CRAN). - Para
empezarytutorialesvisite nuestra página Wiki. - Enlace a un tutorial en video para análisis CITE-Seq y scRNA-Seq: Video
- Todo lo que necesita saber sobre el mapa KNetL: Video
- Si usa
FlowJooSeqGeq, ofrecen plugins para iCellR y otras herramientas de análisis unicelular. Puede encontrar la lista completa de plugins aquí: https://www.flowjo.com/exchange/#/ . Específicamente, el plugin iCellR está aquí: https://www.flowjo.com/exchange/#/plugin/profile?id=34. Además, hay un tutorial de Expresión Diferencial (DE) para SeqGeq disponible para guiarlo en el proceso: Tutorial DE SeqGeq
citar iCellR use este PMID: 34353854iCellR publications: PMID: 35660135 (scRNA-seq/KNetL) PMID: 35180378 (CITE-seq/KNetL), PMID: 34911733 (i.score and cell ranking), PMID: 3496305501427-6?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0092867421014276%3Fshowall%3Dtrue) (scRNA-seq), PMID 31744829 (scRNA-seq), PMID: 31934613 (bulk RNA-seq from TCGA), PMID: 32550269 (scVDJ-seq), PMID: 34135081, PMID: 33593073, PMID: 34634466, PMID: 35302059, PMID: 34353854
Single (i) Cell R package (iCellR)






*
For getting started and tutorials go to our Wiki page.
--- Tranlated By Open Ai Tx | Last indexed: 2025-12-16 ---
