
通过潜在扩散和先验知识提升时空疾病进展模型
Lemuel Puglisi, Daniel C. Alexander, Daniele Ravì
https://github.com/user-attachments/assets/28ad3693-5e3e-4f6e-9bbc-485424fbbee2
新闻
- 🎉 BrLP 荣获 MICCAI 2025 媒体最佳论文奖亚军!
- 🎉 范德堡大学的一篇新论文在BLSA数据集上复现了我们的结果!
- 🆕 BrLP 命令行工具的简明指南已发布!
- 🎉 BrLP 入围并进入 MICCAI 最佳论文奖决选名单!(前 <1%)
- 🎉 BrLP 已被提前接收并入选于 MICCAI 2024的口头报告!(前 4%)
目录
安装
下载仓库,进入项目文件夹(cd),并安装 brlp 包:
pip install -e .数据准备
请查看我们的文档 数据准备与研究可重复性。该文件将指导您如何组织数据并创建运行训练流程所需的 CSV 文件。
训练
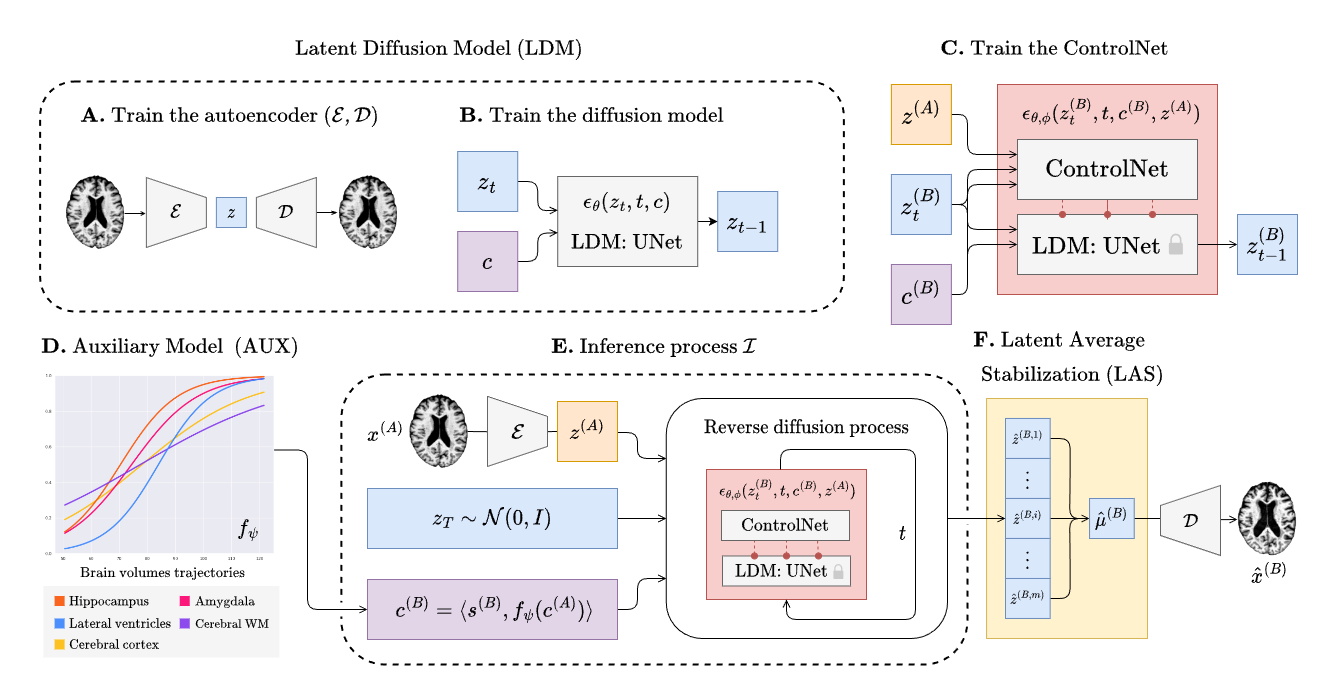
BrLP 训练分为三个主要阶段,后续章节将详细描述。每次训练(辅助模型除外)均可通过 tensorboard 进行监控,方法如下:
tensorboard --logdir runsTrain the autoencoder
Follow the commands below to train the autoencoder.
# Create an output and a cache directory
mkdir ae_output ae_cacheRun the training script
python scripts/training/train_autoencoder.py \
--dataset_csv /path/to/A.csv \
--cache_dir ./ae_cache \
--output_dir ./ae_output然后从您的MRI数据中提取潜变量:
python scripts/prepare/extract_latents.py \
--dataset_csv /path/to/A.csv \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth将 XXX 替换为您选择的自动编码器检查点。
训练 UNet
按照以下命令训练扩散 UNet。将 XXX 替换为您选择的自动编码器检查点。
# Create an output and a cache directory:
mkdir unet_output unet_cacheRun the training script
python scripts/training/train_diffusion_unet.py \
--dataset_csv /path/to/A.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth训练 ControlNet
按照以下命令训练 ControlNet。将 XXX 替换为您选择的自动编码器和 UNet 检查点。
# Create an output and a cache directory:
mkdir cnet_output cnet_cacheRun the training script
python scripts/training/train_controlnet.py \
--dataset_csv /path/to/B.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth \
--diff_ckpt unet_output/unet-ep-XXX.pth辅助模型
按照以下命令训练 DCM 辅助模型。
# Create an output directory
mkdir aux_outputRun the training script
python scripts/training/train_aux.py \
--dataset_csv /path/to/A.csv \
--output_path aux_output推断
我们的软件包附带一个 brlp 命令,用于使用 BrLP 进行推断。请查看:
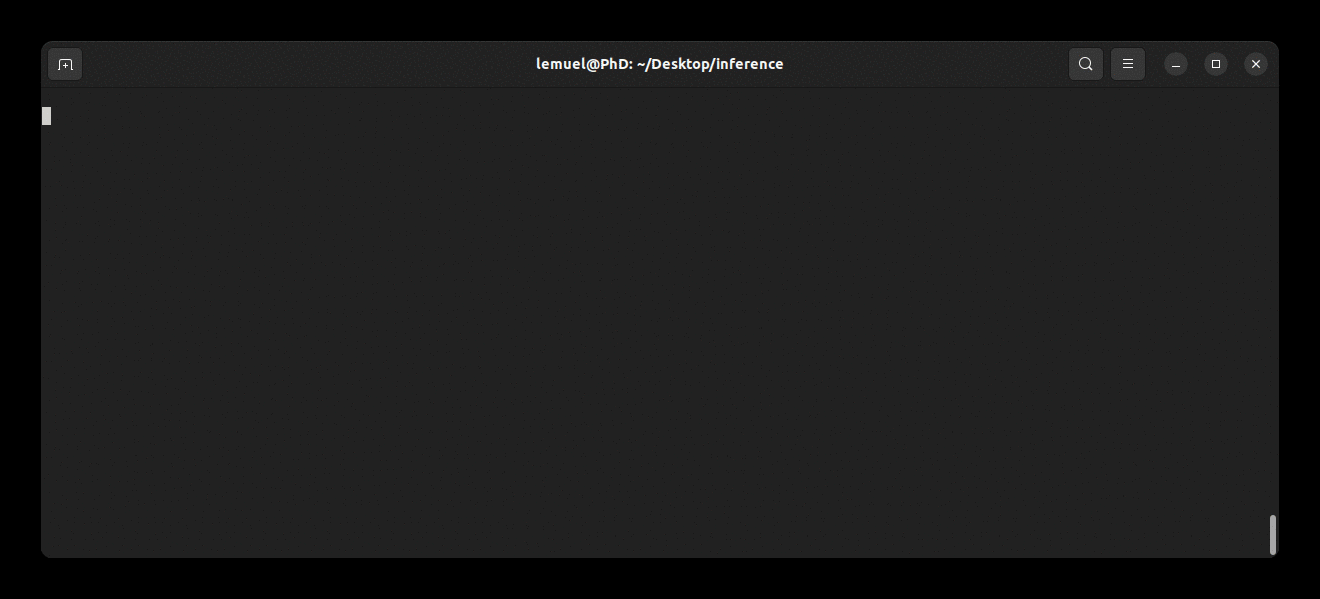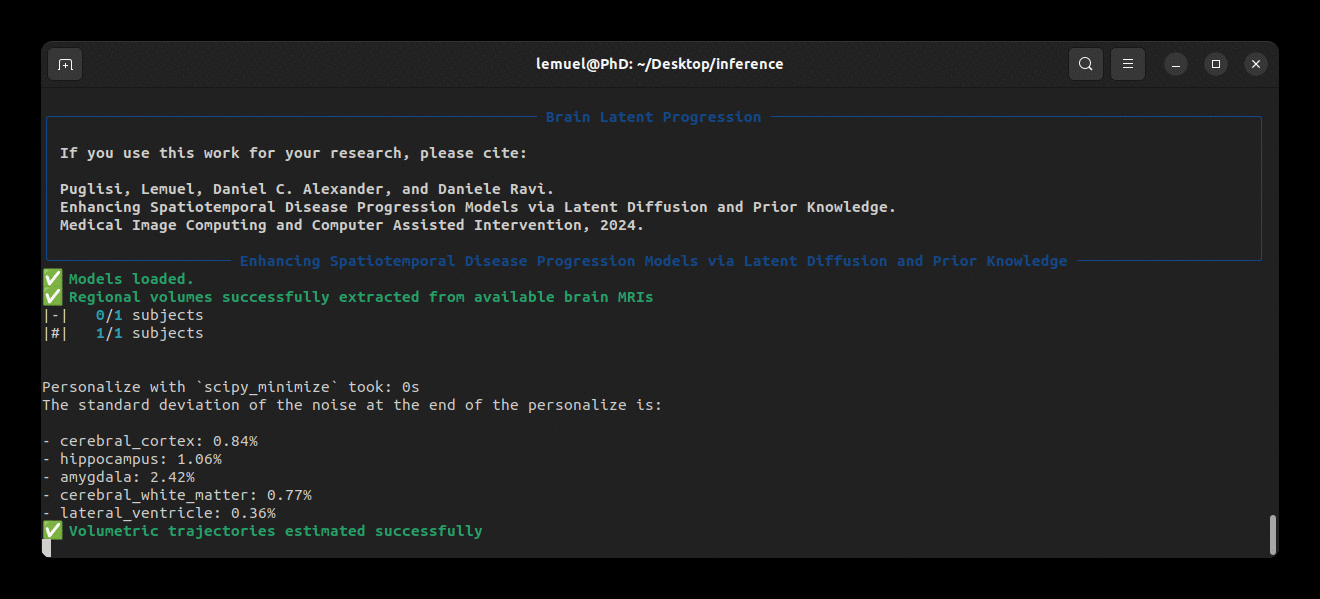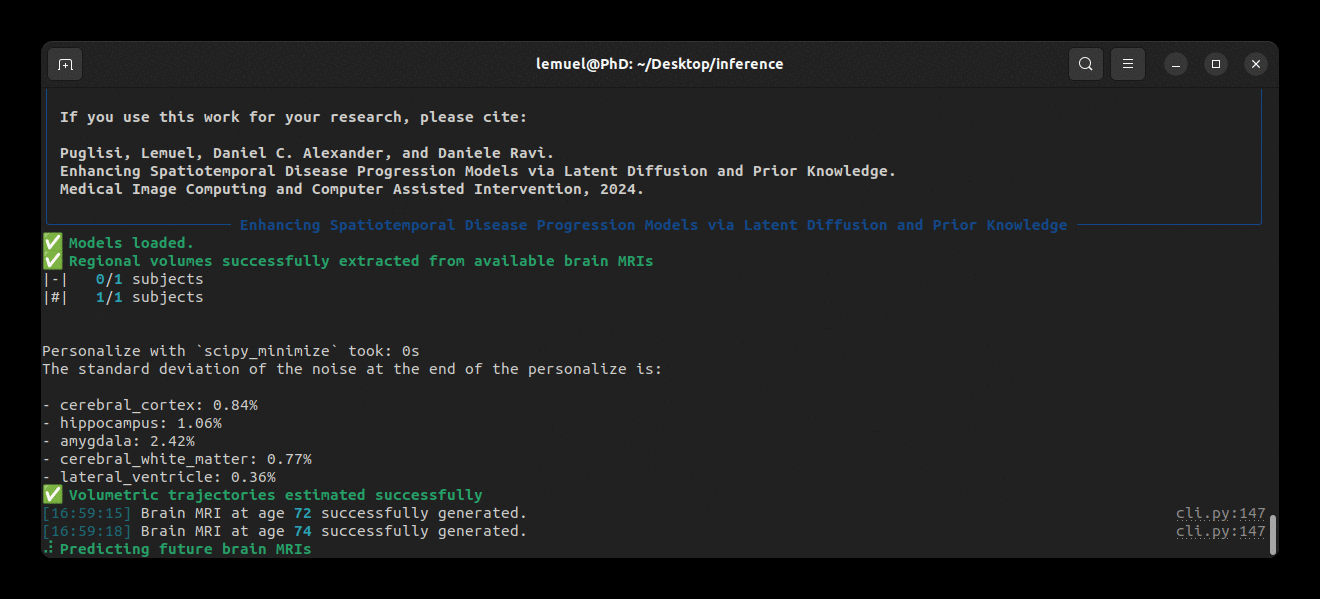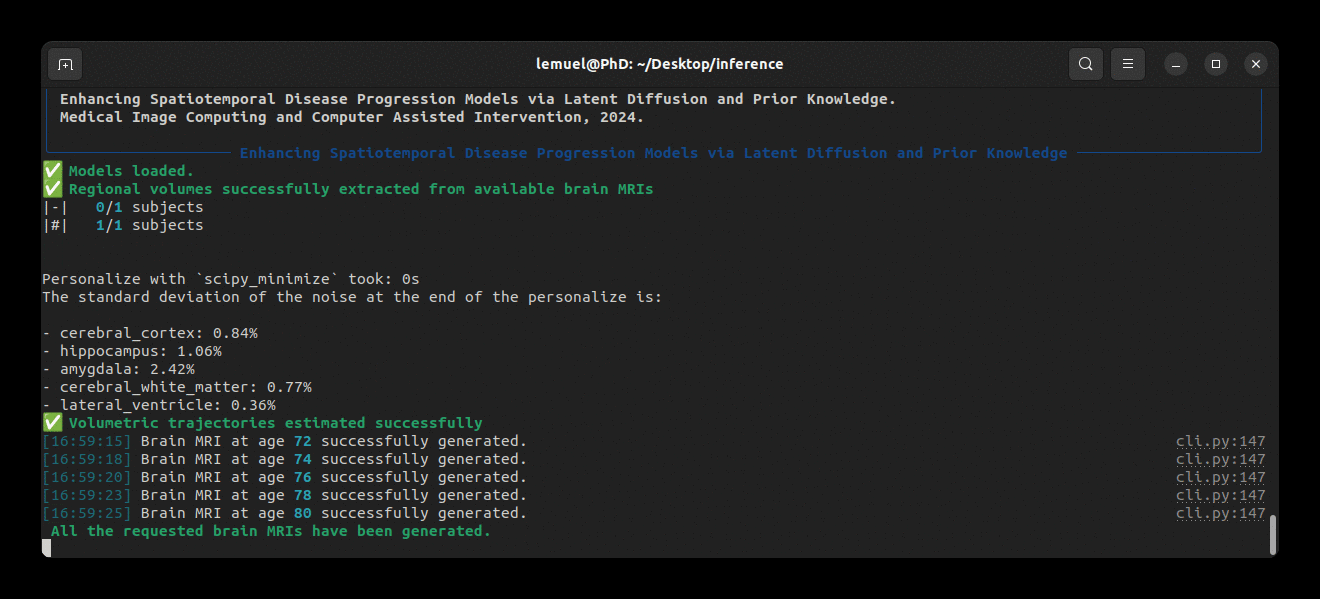
brlp --help--input 参数需要一个 CSV 文件,列出所有受试者的可用数据。示例请查看 examples/input.example.csv。如果你还没有对输入扫描进行分割,brlp 可以使用 SynthSeg 为你执行此任务,但这要求安装 FreeSurfer >= 7.4。--confs 参数指定模型路径及其他推理参数,例如 LAS $m$。示例请查看 examples/confs.example.yaml。程序运行示例如下:
预训练模型
下载 BrLP 的预训练模型:
| 模型 | 权重链接 | | ---------------------- | ------------------------------------------------------------ | | 自编码器 | 链接 | | 扩散模型 UNet | 链接 | | ControlNet | 链接 | | 辅助模型 (DCM) | 链接 |
致谢
感谢开源库维护者们为加速研究进程所做的贡献,特别感谢 MONAI 及其 GenerativeModels 扩展。
引用
医学图像分析:
@article{puglisi2025brain,
title={Brain latent progression: Individual-based spatiotemporal disease progression on 3D brain MRIs via latent diffusion},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
journal={Medical Image Analysis},
year={2025}
}MICCAI 2024 会议论文集:
@inproceedings{puglisi2024enhancing,
title={Enhancing spatiotemporal disease progression models via latent diffusion and prior knowledge},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={173--183},
year={2024},
organization={Springer}
}SPIE 医学成像 2025 会议论文集:
@inproceedings{mcmaster2025technical,
title={A technical assessment of latent diffusion for Alzheimer's disease progression},
author={McMaster, Elyssa and Puglisi, Lemuel and Gao, Chenyu and Krishnan, Aravind R and Saunders, Adam M and Ravi, Daniele and Beason-Held, Lori L and Resnick, Susan M and Zuo, Lianrui and Moyer, Daniel and others},
booktitle={Medical Imaging 2025: Image Processing},
volume={13406},
pages={505--513},
year={2025},
organization={SPIE}
}--- Tranlated By Open Ai Tx | Last indexed: 2026-01-20 ---


