
潜在拡散と事前知識を用いた時空間的疾患進行モデルの強化
レミュエル・プグリジ, ダニエル・C・アレキサンダー, ダニエレ・ラヴィ
https://github.com/user-attachments/assets/28ad3693-5e3e-4f6e-9bbc-485424fbbee2
インストール • トレーニング • CLIアプリケーション • 引用方法
ニュース
- 🎉 BrLPはMICCAI 2025のメディア最優秀論文賞で準優勝を獲得しました!
- 🎉 バンダービルト大学からの新しい論文がBLSAデータセットで私たちの結果を再現しました!
- 🆕 BrLP CLIの簡単ガイドが公開されました!
- 🎉 BrLPはMICCAI最優秀論文賞にノミネートされ、ファイナリストに選出されました!(トップ<1%)
- 🎉 BrLPはMICCAI 2024で早期採択され、口頭発表に選ばれました!(トップ4%)
目次
インストール
リポジトリをダウンロードし、プロジェクトフォルダにcdしてからbrlpパッケージをインストールしてください:
pip install -e .データ準備
データ準備と研究の再現性に関するドキュメントをご覧ください。このファイルは、データの整理方法およびトレーニングパイプラインを実行するために必要なCSVファイルの作成方法を案内します。
トレーニング
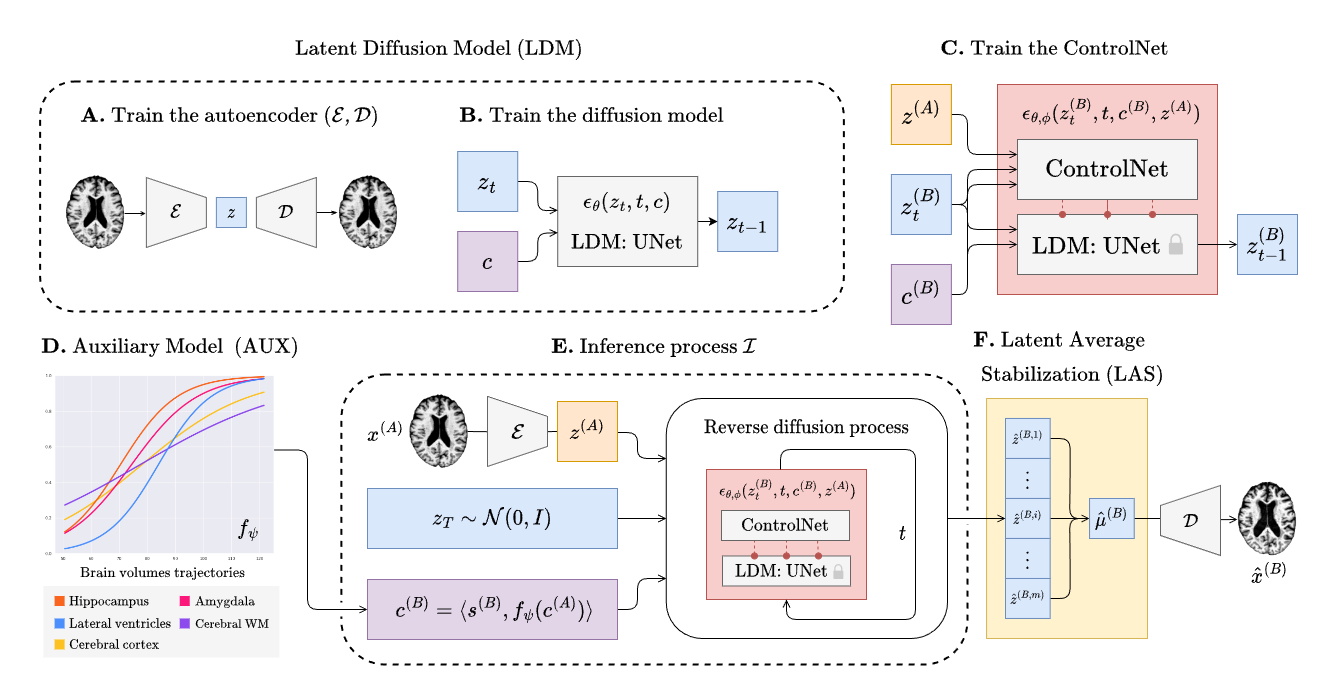
BrLPのトレーニングは、後続のセクションで説明する3つの主要なフェーズがあります。補助モデルを除くすべてのトレーニングは、以下のように tensorboard を使用して監視できます:
tensorboard --logdir runsTrain the autoencoder
Follow the commands below to train the autoencoder.
# Create an output and a cache directory
mkdir ae_output ae_cacheRun the training script
python scripts/training/train_autoencoder.py \
--dataset_csv /path/to/A.csv \
--cache_dir ./ae_cache \
--output_dir ./ae_output次に、MRIデータから潜在変数を抽出します:
python scripts/prepare/extract_latents.py \
--dataset_csv /path/to/A.csv \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthXXX を置き換えて、お好みのオートエンコーダーチェックポイントを選択してください。
UNetのトレーニング
以下のコマンドに従って、拡散UNetをトレーニングします。XXX を置き換えて、お好みのオートエンコーダーチェックポイントを選択してください。
# Create an output and a cache directory:
mkdir unet_output unet_cacheRun the training script
python scripts/training/train_diffusion_unet.py \
--dataset_csv /path/to/A.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthControlNetをトレーニングする
以下のコマンドに従ってControlNetをトレーニングしてください。XXXを選択したオートエンコーダーとUNetのチェックポイントに置き換えてください。
# Create an output and a cache directory:
mkdir cnet_output cnet_cacheRun the training script
python scripts/training/train_controlnet.py \
--dataset_csv /path/to/B.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth \
--diff_ckpt unet_output/unet-ep-XXX.pth補助モデル
以下のコマンドに従ってDCM補助モデルを訓練してください。
# Create an output directory
mkdir aux_outputRun the training script
python scripts/training/train_aux.py \
--dataset_csv /path/to/A.csv \
--output_path aux_output私たちは、時間経過に伴う体積変化を予測できる任意の疾患進行モデルが、BrLPの補助モデルとしても有効であることを強調します。
推論
当パッケージには、BrLPを推論に使用するための brlp コマンドが付属しています。確認してください:
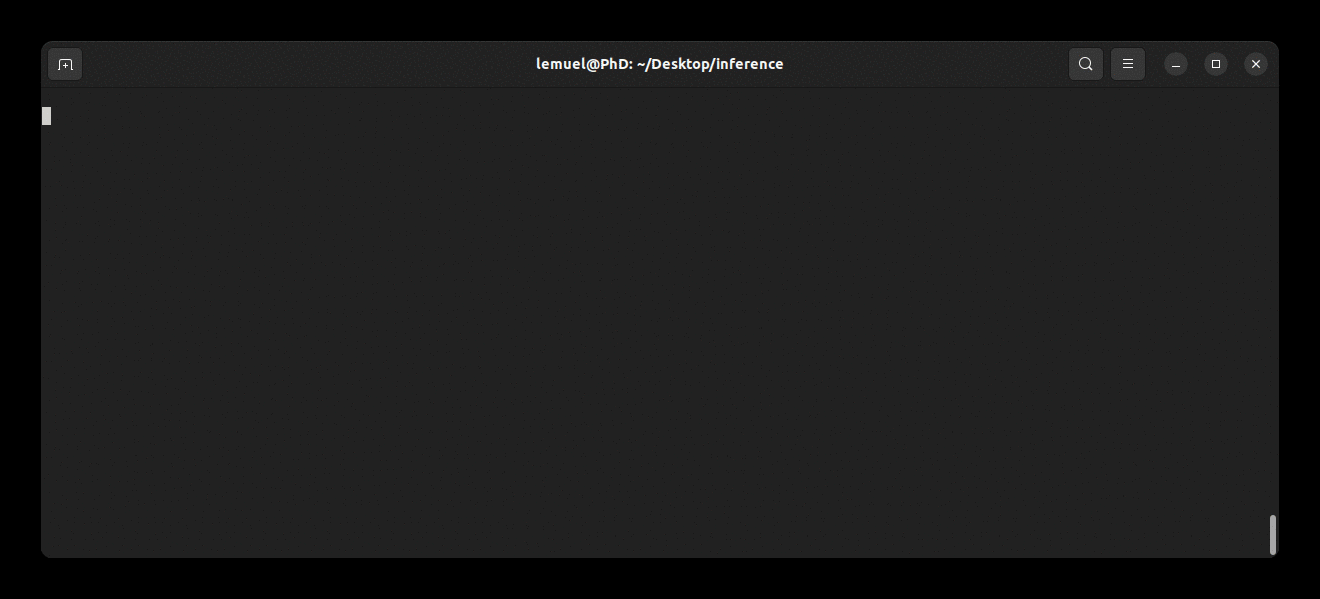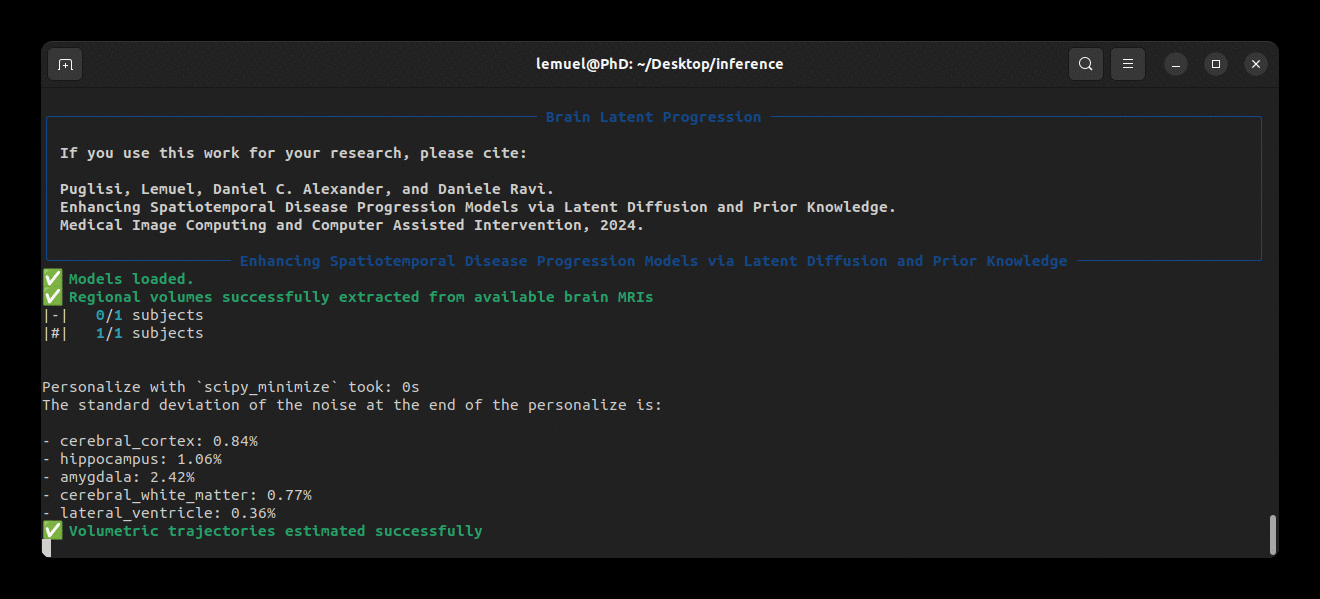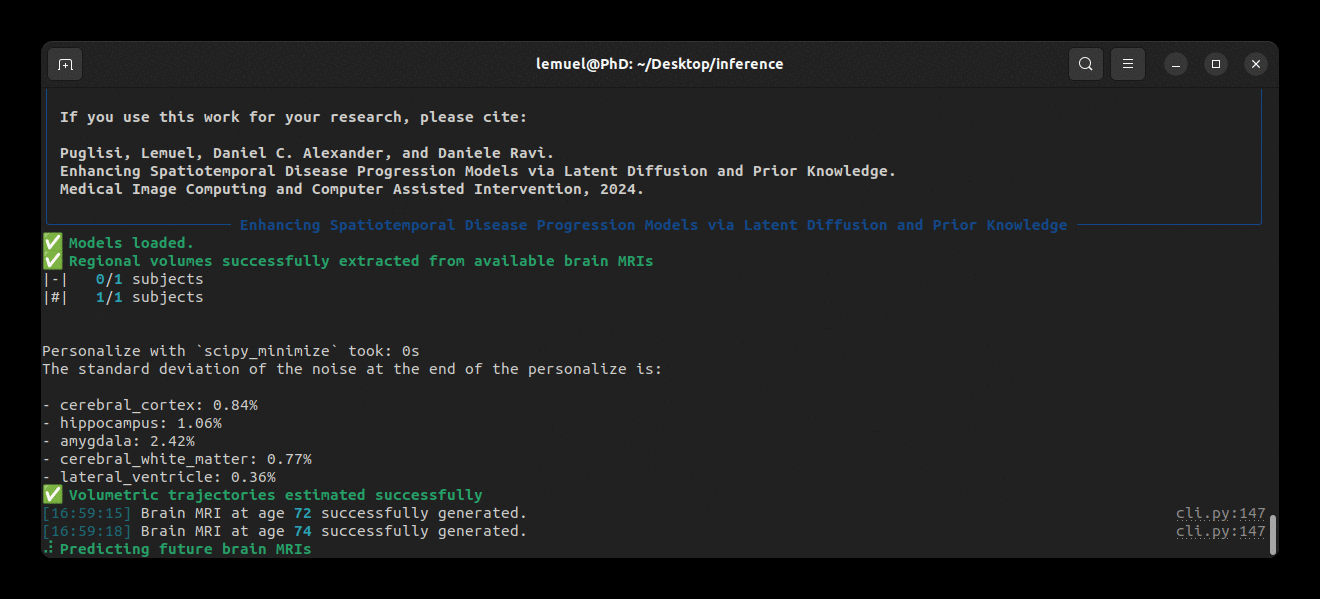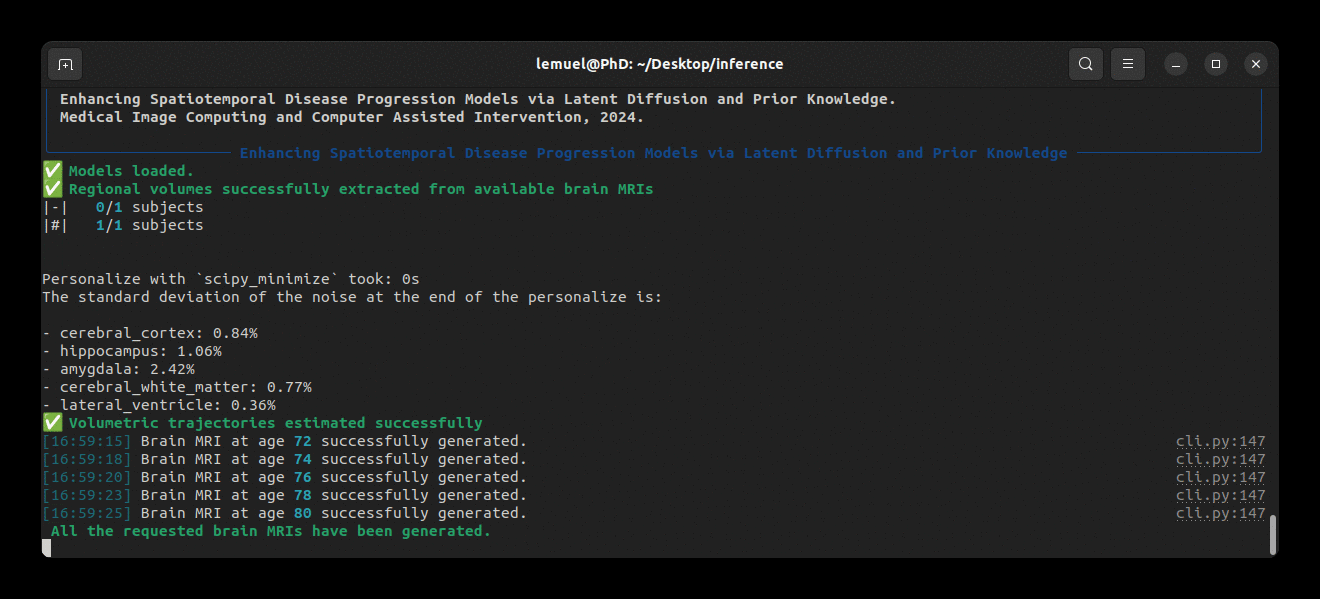
brlp --help--input パラメータは、被験者の利用可能なすべてのデータを一覧にしたCSVファイルを必要とします。例については、examples/input.example.csv を参照してください。入力スキャンのセグメンテーションを行っていない場合、brlp は SynthSeg を使用してこの作業を実行できますが、FreeSurfer >= 7.4 のインストールが必要です。--confs パラメータはモデルへのパスやLAS $m$ などの他の推論パラメータを指定します。例については examples/confs.example.yaml を参照してください。プログラムの実行例は以下の通りです:
事前学習済みモデル
BrLP用の事前学習済みモデルをダウンロードしてください:
| モデル | 重みのURL | | ---------------------- | ------------------------------------------------------------ | | オートエンコーダー | リンク | | ディフュージョンモデルUNet | リンク | | コントロールネット | リンク | | 補助モデル (DCM) | リンク |
謝辞
研究プロセスの加速に貢献しているオープンソースライブラリのメンテナに感謝します。特に MONAI とその GenerativeModels 拡張に特別な謝辞を捧げます。
引用
Medical Image Analysis:
@article{puglisi2025brain,
title={Brain latent progression: Individual-based spatiotemporal disease progression on 3D brain MRIs via latent diffusion},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
journal={Medical Image Analysis},
year={2025}
}MICCAI 2024 論文集:
@inproceedings{puglisi2024enhancing,
title={Enhancing spatiotemporal disease progression models via latent diffusion and prior knowledge},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={173--183},
year={2024},
organization={Springer}
}SPIE メディカルイメージング 2025 論文集:
@inproceedings{mcmaster2025technical,
title={A technical assessment of latent diffusion for Alzheimer's disease progression},
author={McMaster, Elyssa and Puglisi, Lemuel and Gao, Chenyu and Krishnan, Aravind R and Saunders, Adam M and Ravi, Daniele and Beason-Held, Lori L and Resnick, Susan M and Zuo, Lianrui and Moyer, Daniel and others},
booktitle={Medical Imaging 2025: Image Processing},
volume={13406},
pages={505--513},
year={2025},
organization={SPIE}
}--- Tranlated By Open Ai Tx | Last indexed: 2026-01-20 ---


