
Amélioration des modèles spatiotemporels de progression des maladies via la diffusion latente et les connaissances a priori
Lemuel Puglisi, Daniel C. Alexander, Daniele Ravì
https://github.com/user-attachments/assets/28ad3693-5e3e-4f6e-9bbc-485424fbbee2
Installation • Entraînement • Application CLI • Citer
NOUVELLES
- 🎉 BrLP a obtenu la deuxième place pour le Media Best Paper Award à MICCAI 2025 !
- 🎉 Un nouvel article de l'Université Vanderbilt a reproduit nos résultats sur le jeu de données BLSA !
- 🆕 Le guide court sur l'utilisation du CLI BrLP est disponible !
- 🎉 BrLP a été nommé et présélectionné pour le MICCAI Best Paper Award ! (top <1%)
- 🎉 BrLP a été accepté en avance et sélectionné pour une présentation orale à MICCAI 2024 (top 4%) !
Table des matières
- Installation
- Préparation des données
- Entraînement
- Modèles préentraînés
- Remerciements
- Citation de notre travail
Installation
Téléchargez le dépôt, faites un cd dans le dossier du projet et installez le paquet brlp :
pip install -e .Préparation des données
Consultez notre document sur la Préparation des données et reproductibilité de l'étude. Ce fichier vous guidera dans l'organisation de vos données et la création des fichiers CSV nécessaires pour exécuter les pipelines d'entraînement.
Entraînement
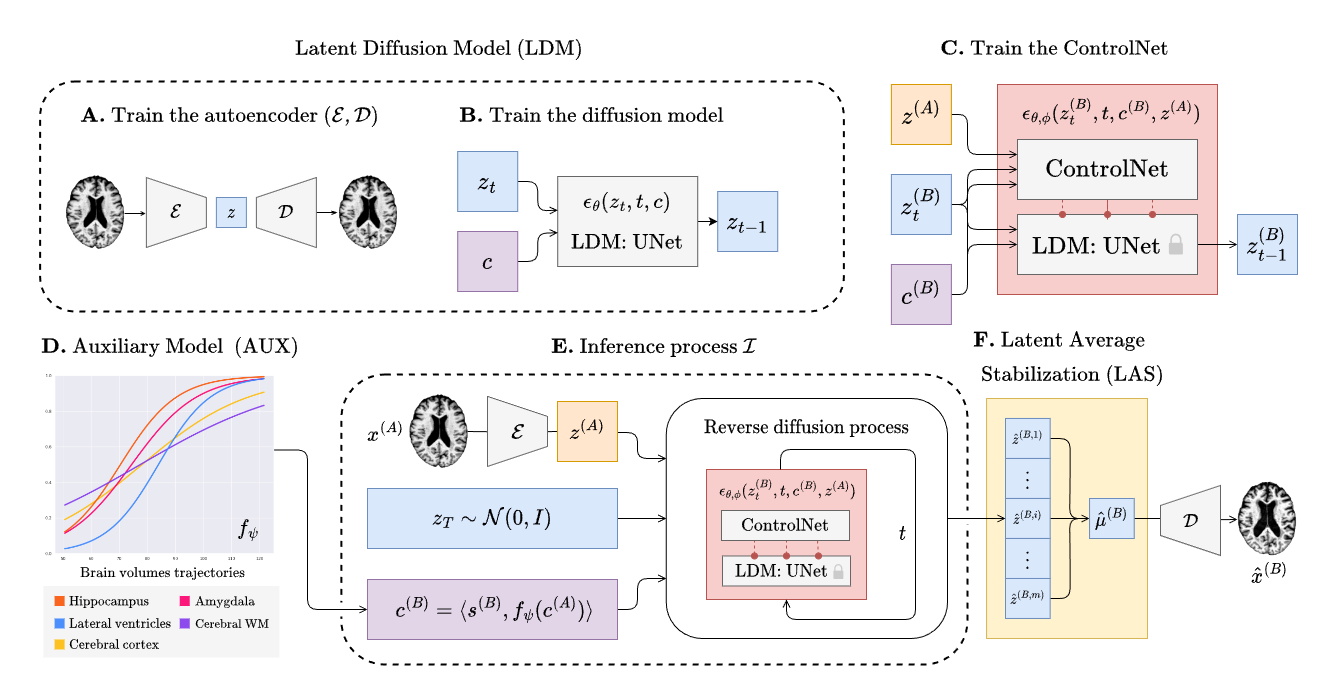
L'entraînement de BrLP comporte 3 phases principales qui seront décrites dans les sections suivantes. Chaque entraînement (sauf pour le modèle auxiliaire) peut être suivi à l'aide de tensorboard comme suit :
tensorboard --logdir runsTrain the autoencoder
Follow the commands below to train the autoencoder.
# Create an output and a cache directory
mkdir ae_output ae_cacheRun the training script
python scripts/training/train_autoencoder.py \
--dataset_csv /path/to/A.csv \
--cache_dir ./ae_cache \
--output_dir ./ae_outputPuis extrayez les latents de vos données IRM :
python scripts/prepare/extract_latents.py \
--dataset_csv /path/to/A.csv \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthRemplacez XXX pour sélectionner les points de contrôle de l'autoencodeur de votre choix.
Entraînez le UNet
Suivez les commandes ci-dessous pour entraîner le UNet de diffusion. Remplacez XXX pour sélectionner les points de contrôle de l'autoencodeur de votre choix.
# Create an output and a cache directory:
mkdir unet_output unet_cacheRun the training script
python scripts/training/train_diffusion_unet.py \
--dataset_csv /path/to/A.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthEntraînez le ControlNet
Suivez les commandes ci-dessous pour entraîner le ControlNet. Remplacez XXX pour sélectionner les points de contrôle de l'autoencodeur et de l'UNet de votre choix.
# Create an output and a cache directory:
mkdir cnet_output cnet_cacheRun the training script
python scripts/training/train_controlnet.py \
--dataset_csv /path/to/B.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth \
--diff_ckpt unet_output/unet-ep-XXX.pthModèles auxiliaires
Suivez les commandes ci-dessous pour entraîner le modèle auxiliaire DCM.
# Create an output directory
mkdir aux_outputRun the training script
python scripts/training/train_aux.py \
--dataset_csv /path/to/A.csv \
--output_path aux_outputNous soulignons que tout modèle de progression de maladie capable de prédire les changements volumétriques au fil du temps est également viable en tant que modèle auxiliaire pour BrLP.
Inférence
Notre package est livré avec une commande brlp pour utiliser BrLP pour l'inférence. Vérifiez :
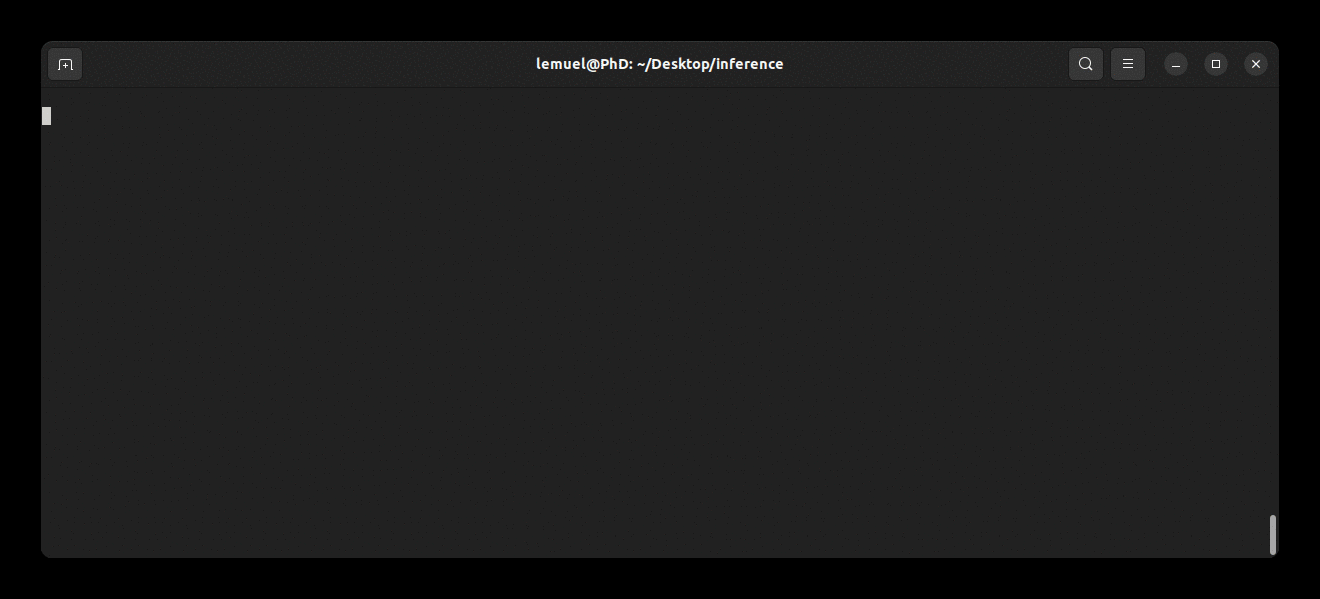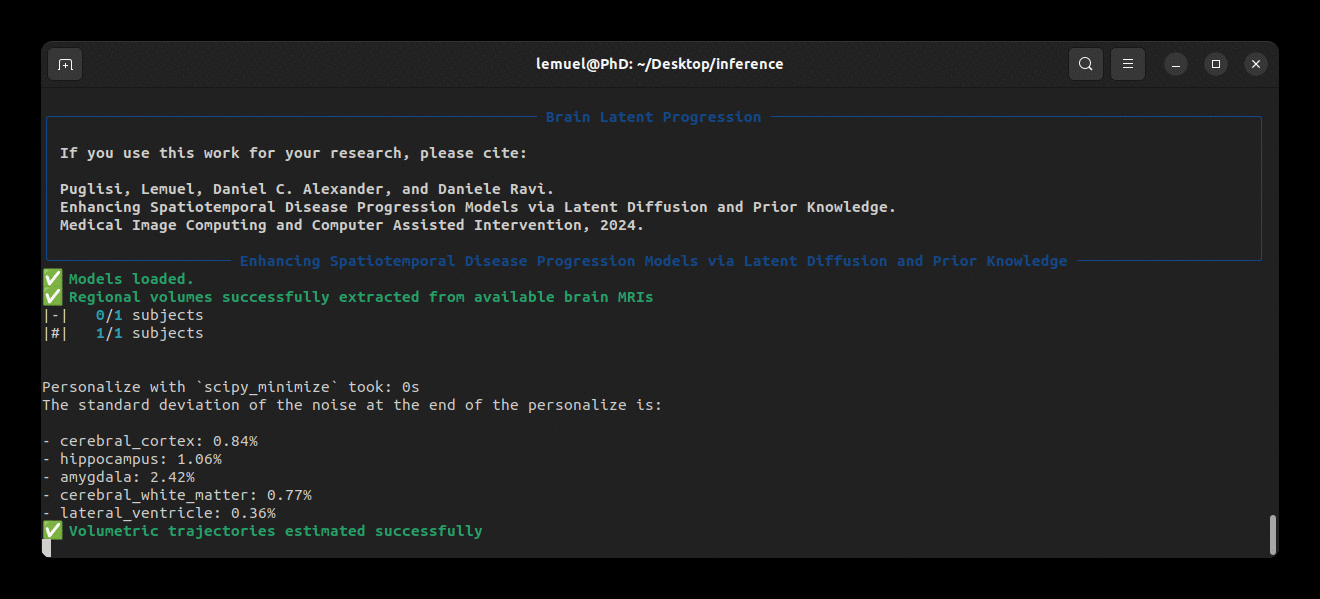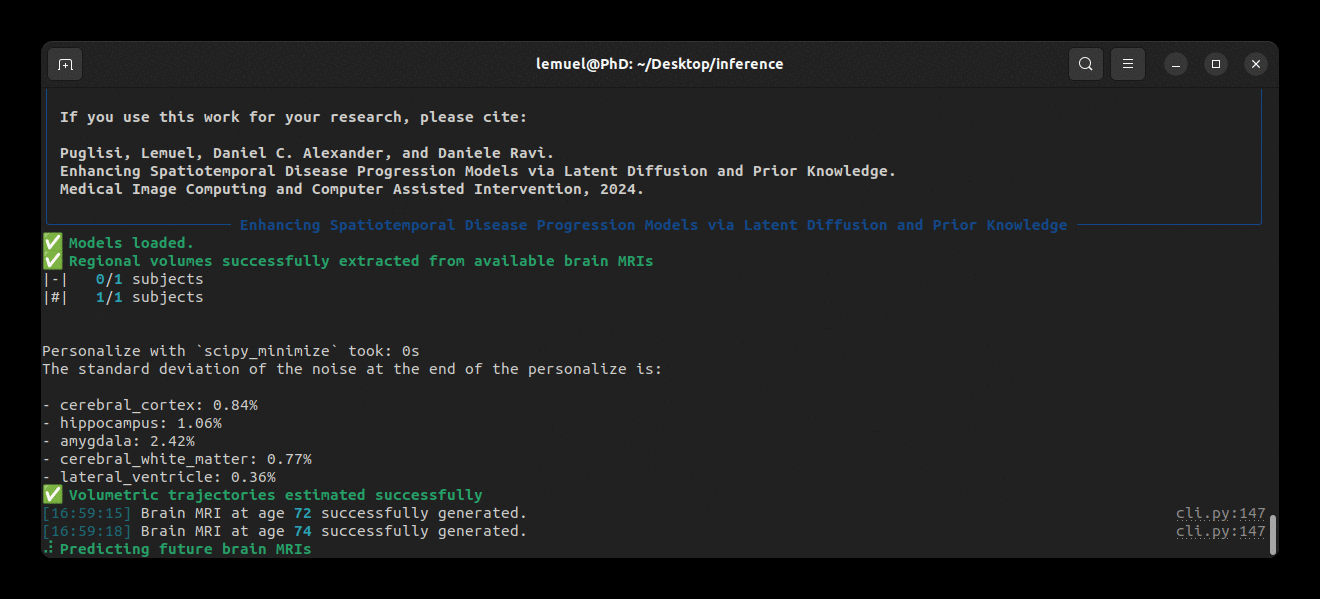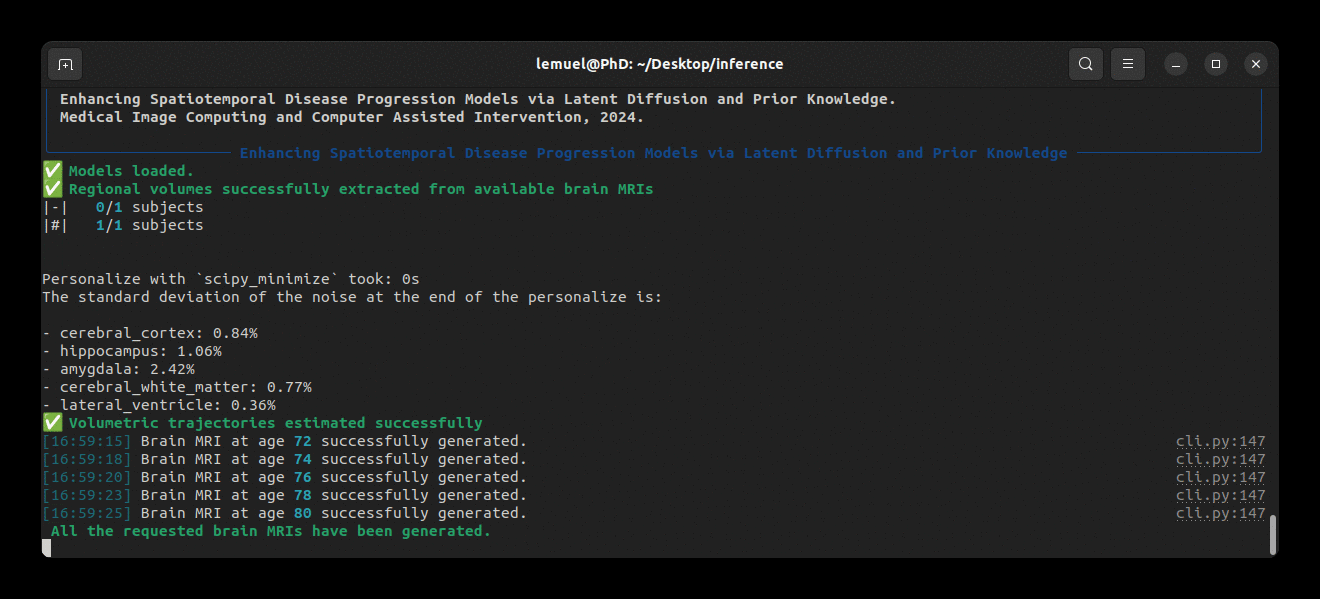
brlp --help--input requiert un fichier CSV dans lequel vous listez toutes les données disponibles pour vos sujets. Pour un exemple, consultez examples/input.example.csv. Si vous n'avez pas segmenté vos scans d'entrée, brlp peut effectuer cette tâche pour vous en utilisant SynthSeg, mais cela nécessite que FreeSurfer >= 7.4 soit installé. Le paramètre --confs spécifie les chemins vers les modèles et autres paramètres d'inférence, tels que LAS $m$. Pour un exemple, consultez examples/confs.example.yaml. L'exécution du programme se présente ainsi :
Modèles pré-entraînés
Téléchargez les modèles pré-entraînés pour BrLP :
| Modèle | URL des poids | | ---------------------- | ------------------------------------------------------------ | | Autoencodeur | lien | | Modèle de diffusion UNet | lien | | ControlNet | lien | | Modèles auxiliaires (DCM) | lien |
Remerciements
Nous remercions les mainteneurs des bibliothèques open-source pour leurs contributions à l'accélération du processus de recherche, avec une mention spéciale à MONAI et son extension GenerativeModels.
Citation
Medical Image Analysis :
@article{puglisi2025brain,
title={Brain latent progression: Individual-based spatiotemporal disease progression on 3D brain MRIs via latent diffusion},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
journal={Medical Image Analysis},
year={2025}
}Actes de MICCAI 2024 :
@inproceedings{puglisi2024enhancing,
title={Enhancing spatiotemporal disease progression models via latent diffusion and prior knowledge},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={173--183},
year={2024},
organization={Springer}
}@inproceedings{mcmaster2025technical,
title={A technical assessment of latent diffusion for Alzheimer's disease progression},
author={McMaster, Elyssa and Puglisi, Lemuel and Gao, Chenyu and Krishnan, Aravind R and Saunders, Adam M and Ravi, Daniele and Beason-Held, Lori L and Resnick, Susan M and Zuo, Lianrui and Moyer, Daniel and others},
booktitle={Medical Imaging 2025: Image Processing},
volume={13406},
pages={505--513},
year={2025},
organization={SPIE}
}--- Tranlated By Open Ai Tx | Last indexed: 2026-01-20 ---


