
Mejorando modelos de progresión de enfermedades espaciotemporales mediante difusión latente y conocimiento previo
Lemuel Puglisi, Daniel C. Alexander, Daniele Ravì
https://github.com/user-attachments/assets/28ad3693-5e3e-4f6e-9bbc-485424fbbee2
Instalación • Entrenamiento • Aplicación CLI • Citar
NOTICIAS
- 🎉 ¡BrLP ha obtenido el segundo lugar en el premio Media Best Paper Award en MICCAI 2025!
- 🎉 Un nuevo artículo de la Universidad de Vanderbilt ha replicado nuestros resultados en el conjunto de datos BLSA!
- 🆕 ¡La guía corta para usar la CLI de BrLP ya está disponible!
- 🎉 ¡BrLP ha sido nominado y preseleccionado para el MICCAI Best Paper Award! (top <1%)
- 🎉 BrLP ha sido aceptado anticipadamente y seleccionado para presentación oral en MICCAI 2024 (top 4%)!
Tabla de Contenidos
- Instalación
- Preparación de datos
- Entrenamiento
- Modelos preentrenados
- Agradecimientos
- Citación de nuestro trabajo
Instalación
Descargue el repositorio, haga cd en la carpeta del proyecto e instale el paquete brlp:
pip install -e .Preparación de datos
Consulta nuestro documento sobre Preparación de datos y reproducibilidad del estudio. Este archivo te guiará en la organización de tus datos y la creación de los archivos CSV necesarios para ejecutar los pipelines de entrenamiento.
Entrenamiento
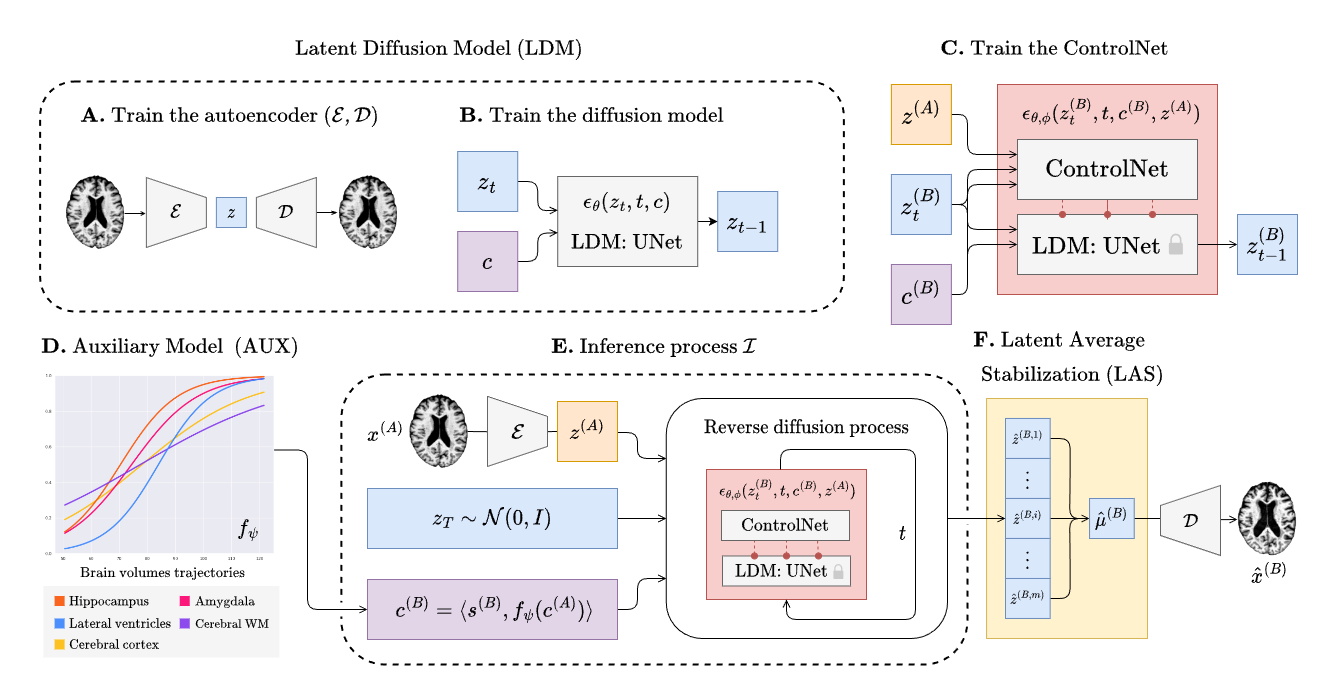
El entrenamiento de BrLP tiene 3 fases principales que se describirán en las secciones siguientes. Cada entrenamiento (excepto para el modelo auxiliar) puede ser monitoreado usando tensorboard de la siguiente manera:
tensorboard --logdir runsTrain the autoencoder
Follow the commands below to train the autoencoder.
# Create an output and a cache directory
mkdir ae_output ae_cacheRun the training script
python scripts/training/train_autoencoder.py \
--dataset_csv /path/to/A.csv \
--cache_dir ./ae_cache \
--output_dir ./ae_outputLuego extrae los latentes de tus datos de MRI:
python scripts/prepare/extract_latents.py \
--dataset_csv /path/to/A.csv \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthReemplace XXX para seleccionar los puntos de control del autoencoder de su elección.
Entrenar el UNet
Siga los comandos a continuación para entrenar el UNet de difusión. Reemplace XXX para seleccionar los puntos de control del autoencoder de su elección.
# Create an output and a cache directory:
mkdir unet_output unet_cacheRun the training script
python scripts/training/train_diffusion_unet.py \
--dataset_csv /path/to/A.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pthEntrena el ControlNet
Sigue los comandos a continuación para entrenar el ControlNet. Reemplaza XXX para seleccionar los puntos de control del autoencoder y UNet de tu elección.
# Create an output and a cache directory:
mkdir cnet_output cnet_cacheRun the training script
python scripts/training/train_controlnet.py \
--dataset_csv /path/to/B.csv \
--cache_dir unet_cache \
--output_dir unet_output \
--aekl_ckpt ae_output/autoencoder-ep-XXX.pth \
--diff_ckpt unet_output/unet-ep-XXX.pthModelos auxiliares
Sigue los comandos a continuación para entrenar el modelo auxiliar DCM.
# Create an output directory
mkdir aux_outputRun the training script
python scripts/training/train_aux.py \
--dataset_csv /path/to/A.csv \
--output_path aux_outputEnfatizamos que cualquier modelo de progresión de la enfermedad capaz de predecir cambios volumétricos a lo largo del tiempo también es viable como modelo auxiliar para BrLP.
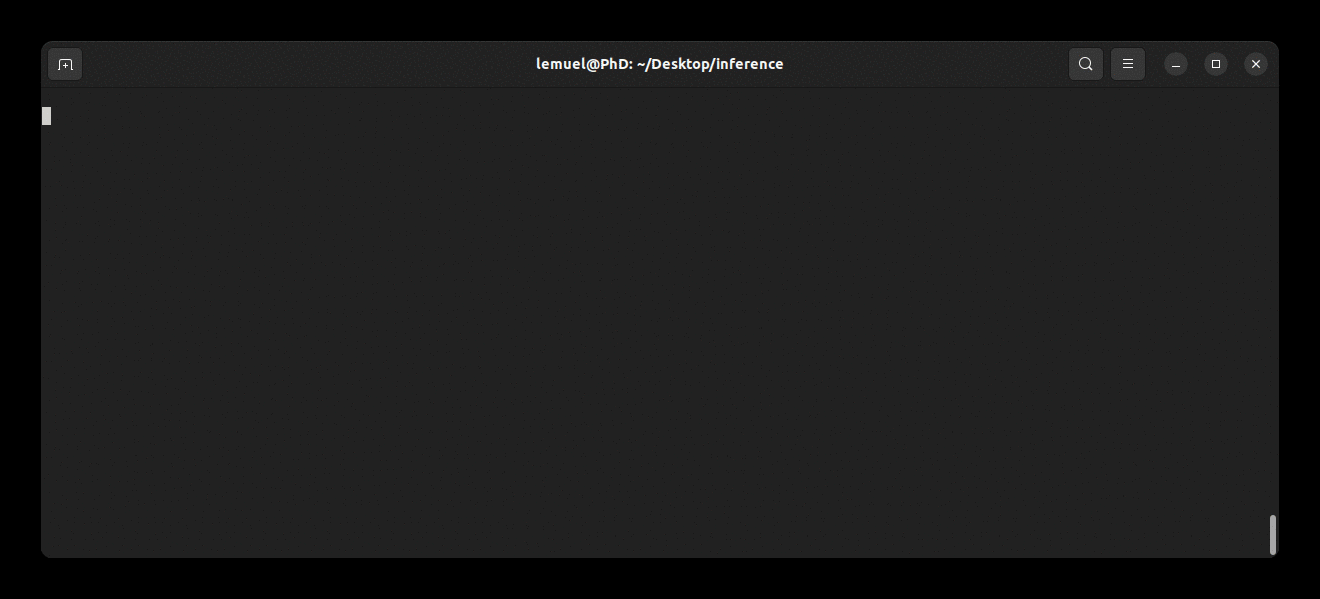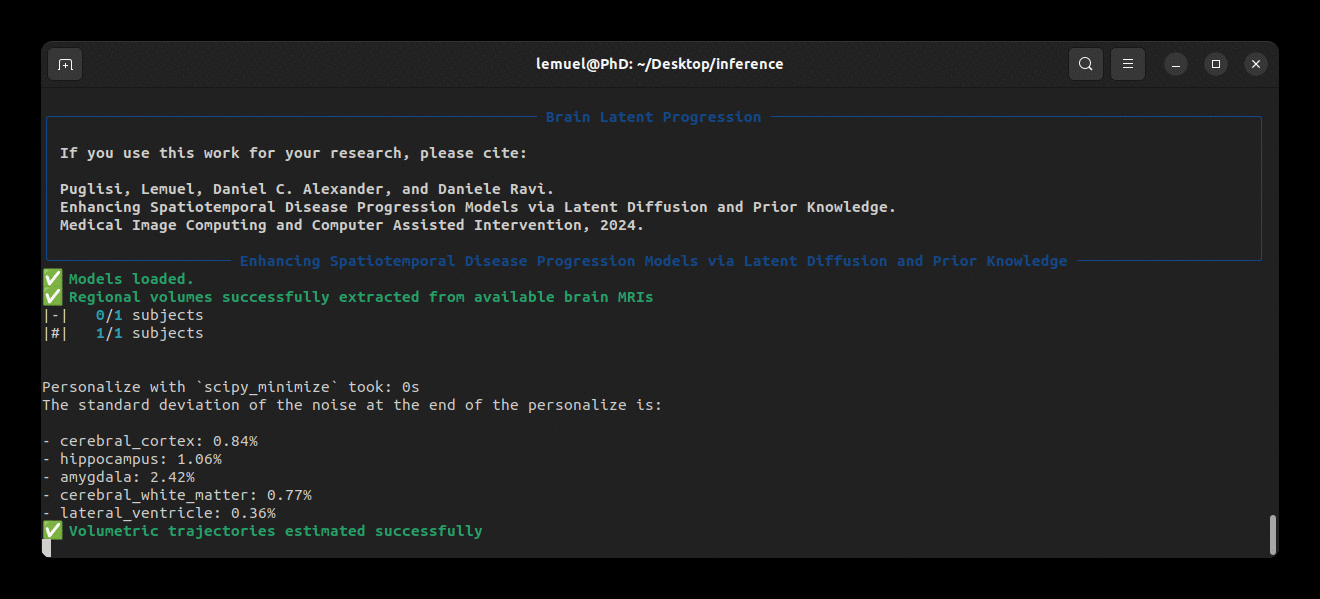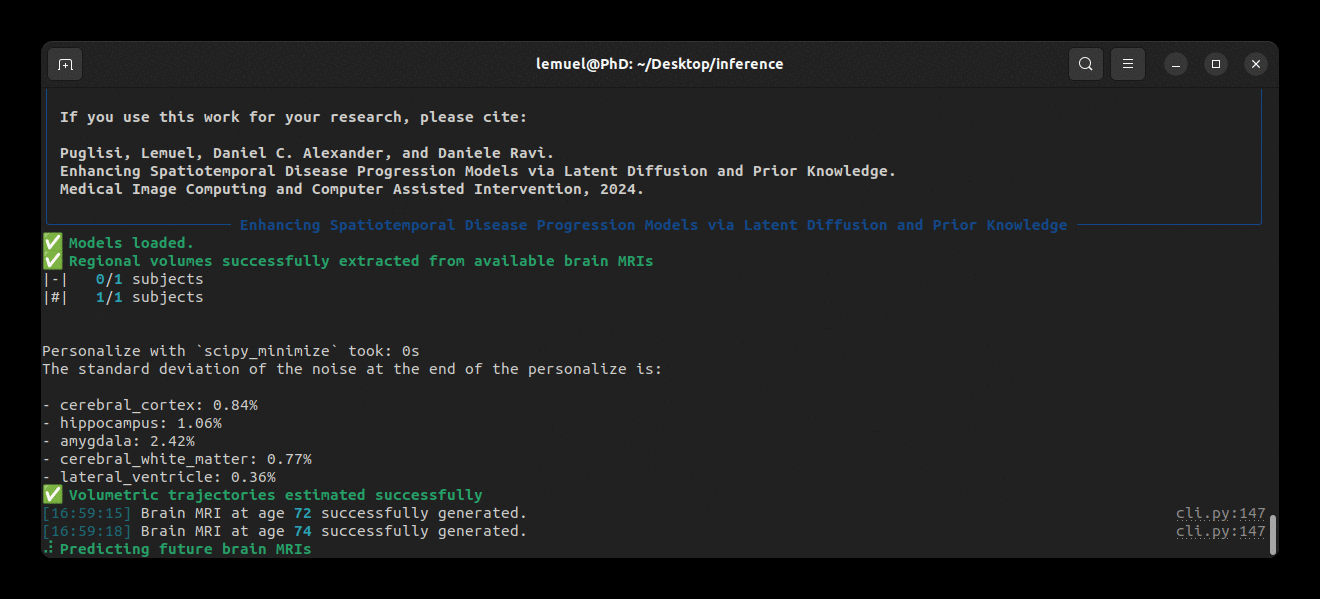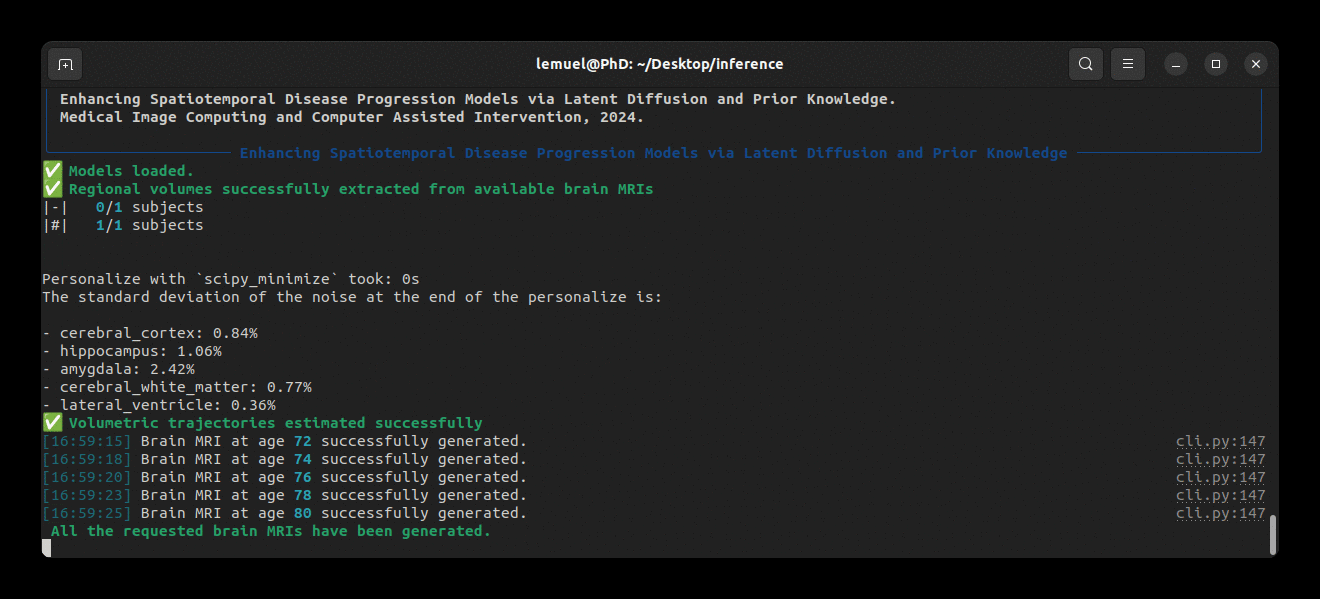
Inferencia
Nuestro paquete incluye un comando brlp para usar BrLP en inferencia. Verifique:
brlp --help--input requiere un archivo CSV donde liste todos los datos disponibles para sus sujetos. Como ejemplo, consulte examples/input.example.csv. Si no ha segmentado sus escaneos de entrada, brlp puede realizar esta tarea por usted usando SynthSeg, pero requiere que FreeSurfer >= 7.4 esté instalado. El parámetro --confs especifica las rutas a los modelos y otros parámetros de inferencia, como LAS $m$. Como ejemplo, consulte examples/confs.example.yaml. Ejecutar el programa se ve así:
Modelos preentrenados
Descargue los modelos preentrenados para BrLP:
| Modelo | URL de Pesos | | ---------------------- | ---------------------------------------------------------- | | Autoencoder | enlace | | Modelo de Difusión UNet| enlace | | ControlNet | enlace | | Modelos Auxiliares (DCM)| enlace |
Agradecimientos
Agradecemos a los mantenedores de bibliotecas de código abierto por sus contribuciones para acelerar el proceso de investigación, con una mención especial a MONAI y su extensión GenerativeModels.
Citación
Medical Image Analysis:
@article{puglisi2025brain,
title={Brain latent progression: Individual-based spatiotemporal disease progression on 3D brain MRIs via latent diffusion},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
journal={Medical Image Analysis},
year={2025}
}Actas de MICCAI 2024:
@inproceedings{puglisi2024enhancing,
title={Enhancing spatiotemporal disease progression models via latent diffusion and prior knowledge},
author={Puglisi, Lemuel and Alexander, Daniel C and Rav{\\i}, Daniele},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={173--183},
year={2024},
organization={Springer}
}Actas de SPIE Medical Imaging 2025:
@inproceedings{mcmaster2025technical,
title={A technical assessment of latent diffusion for Alzheimer's disease progression},
author={McMaster, Elyssa and Puglisi, Lemuel and Gao, Chenyu and Krishnan, Aravind R and Saunders, Adam M and Ravi, Daniele and Beason-Held, Lori L and Resnick, Susan M and Zuo, Lianrui and Moyer, Daniel and others},
booktitle={Medical Imaging 2025: Image Processing},
volume={13406},
pages={505--513},
year={2025},
organization={SPIE}
}--- Tranlated By Open Ai Tx | Last indexed: 2026-01-20 ---


